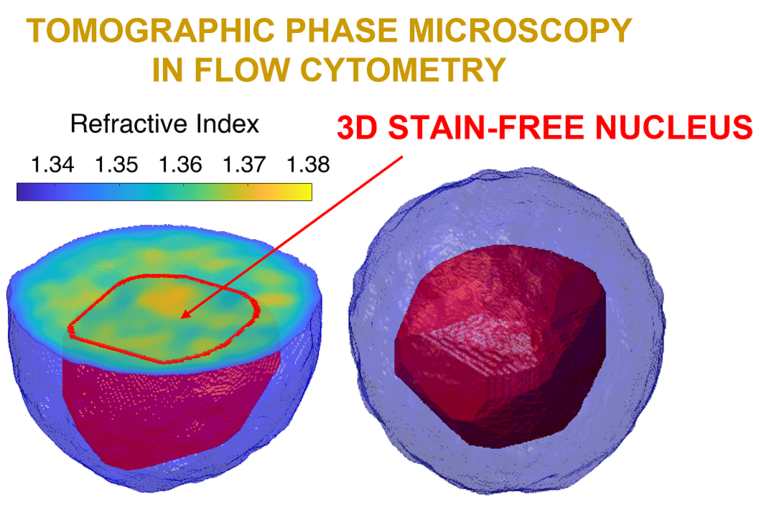
È pronta la ‘Tac’ per singole cellule: si tratta di una tecnica innovativa che, ricreando la loro struttura in 3D, permette di studiare le cellule e distinguere quelle sane da quelle malate, e che promette di aprire la strada ad una svolta nella diagnosi precoce dei tumori e nella sperimentazione di nuovi farmaci. La tecnica è stata messa a punto da un gruppo internazionale di ricercatori guidati da Istituto di Scienze Applicate e Sistemi Intelligenti ‘E. Caianiello’ del Consiglio Nazionale delle Ricerche (Cnr-Isasi), Università Federico II di Napoli e Ceinge - Biotecnologie Avanzate Franco Salvatore, insieme al Politecnico Federale svizzero di Losanna. Lo studio è stato pubblicato sulla rivista Nature Photonics.
I ricercatori, guidati da Daniele Pirone, sono riusciti ad individuare e visualizzare in 3D il nucleo di cellule tumorali senza utilizzare coloranti chimici, che limitano le attuali tecniche richiedendo molto tempo ed alterando le proprietà cellulari. Alla base dei risultati ottenuti c’è una tecnologia di olografia digitale, che consente di analizzare le singole cellule praticamente in tempo reale, ricostruendone l’immagine tridimensionale con una accuratezza senza precedenti. Secondo gli autori dello studio, la tecnica aprirà strade completamente nuove per gli studi di biologia cellulare.
“L’applicazione più promettente è la biopsia liquida, l’indagine meno invasiva attualmente praticabile, che mira alla diagnosi precoce dei tumori e alla definizione di terapie oncologiche personalizzate”, spiega Achille Iolascon di Ceinge e Università Federico II, uno degli autori dello studio. “La tecnica messa a punto in questo studio permetterà di sviluppare metodologie in grado di monitorare ed estrarre le cellule tumorali che circolano nel sangue del paziente”.
Riproduzione riservata © Copyright ANSA